Catherine A. Ball, Erin Battat, Jake K. Byrnes, Peter Carbonetto, Kenneth G. Chahine, Ross E. Curtis, Eyal Elyashiv, Ahna Girshick, Julie M. Granka, Harendra Guturu, Eunjung Han, Ariel Hippen Anderson, Eurie Hong, Amir Kermany, Natalie M. Myres, Keith Noto, Kristin A. Rand, Shiya Song, Yong Wang(in alphabetischer Reihenfolge).
1. Einleitung
AncestryDNA™ bietet genetische Analysen, um Kunden dabei zu helfen, Ihre Familiengeschichte zu entdecken, zu erhalten und zu teilen. Einige der bis dato angebotenen Funktionalitäten basieren exklusiv auf genetischen Informationen. Diese schließen eine genetische Herkunfts- oder Abstammungsschlussfolgerung (wie im Whitepaper: Einschätzung der genetischen Herkunft) und eine identity-by-descent (IBD) oder DNA-Analyse (Whitepaper: Genetische Übereinstimmung). Andere Funktionen, wie DNA Circles, greifen auf die Integration von Abstammungs- und IBD-Daten innerhalb der gesamten AncestryDNA-Datenbank zurück (Whitepaper: DNA Circles). Jede dieser Funktionalitäten bieten einem Kunden ergänzende Informationen: (1) der Abstammungsmix zeigt ein entferntes Bild der genetischen Herkunft eines Kunden, möglicherweise hunderte oder tausende Jahre zurückliegend; (2) DNA-Übereinstimmungen bieten dem Kunden eine Liste von anderen AncestryDNA-Testern, die eine Verwandtschaft aufweisen und mit denen sie oder er einen gemeinsamen Vorfahren innerhalb der letzten 10 Generationen teilt; (3) DNA Circle integrieren IBD- und Stammbaumdaten, um dem Kunden Verwandtschaftsgruppen zu bieten, die augenscheinlich aufgrund eines gemeinsamen Vorfahren DNA miteinander teilen, und so ihre Verbindung zu diesem Vorfahren potenziell verstärken. In Kombination bieten diese Funktionen ein detailliertes Bild der genetischen Abstammung einer Person.
Hier ergänzen wir diese DNA- und stammbaumbasierten Einblicke weiter mit unserer neuen Funktion „Bevölkerungsgruppen“ (Abbildung 1.1). Anstatt die IBD-Verbindungen zwischen jedem einzelnen Kundenpaar in Betracht zu ziehen, analysieren wir simultan mehr als 20 Milliarden Verbindungen, die bei über 2 Millionen AncestryDNA Mitgliedern als großes genetisches Netzwerk (Beschreibung unten in Abschnitt 3) identifiziert wurden. Da die geschätzten IBD-Verbindungen zwischen Personen wahrscheinlich aufgrund einer relativ modernen gemeinsamen Herkunft (innerhalb der vergangenen 10 Generationen) auftreten, repräsentieren breitere Muster in diesem großen Netzwerk wahrscheinlich eine jüngere geteilte Geschichte. Dadurch können wir anhand spezifischer bzw. jüngerer geteilter Geschichte, Cluster von lebenden Personen identifizieren, die einen DNA-Anteil miteinander teilen. Beispielsweise identifizieren wir Gruppen von Kunden, die wahrscheinlich von Immigranten einer bestimmten Migrationswelle abstammen (z. B. Iren, die der Großen Hungersnot entfliehen) oder Kunden, die von Ureinwohnern abstammen, die viele Generationen am gleichen geografischen Ort verbracht haben (z B. frühe Siedler der Appalachen). Nach der Identifizierung dieser Personen-Cluster im gesamten Netzwerk, können wir alle AncestryDNA Kunden einem oder mehreren dieser Cluster auf Grundlage ihres IBD mit anderen AncestryDNA Mitgliedern zuweisen. Diese Zuweisungen können einem Kunden Einblicke in ihre Herkunftsgeschichte bieten, in einigen Fällen können diese sogar auf historische Ereignisse zurückgeführt werden.
In den folgenden Abschnitten beschreiben wir die wissenschaftlichen Prinzipien hinter dem genetischen Netzwerk (Abschnitte 2 und 3), wie wir Cluster darin identifizieren (Abschnitte 4 und 6), wie wir DNA- und Stammbaumdaten verwenden, um diese Cluster zu annotieren (Abschnitt 5) und zu guter Letzt, unsere Methode zur Zuweisung von Kundenproben zu diesen Clustern (Abschnitt 7).
.jpg)
2. Motivation der Populationsgenetik für Bevölkerungsgruppen
In diesem Abschnitt diskutieren wir einige grundlegende Konzepte der Populationsgenetik, die die Identifizierung von Bevölkerungsgruppen anregen und mit einem Beispiel abschließen.
Zuerst stellen wir einige Fachbegriffe vor. Ein IBD-Netzwerk ist eine Repräsentation der genetischen Verbindungen innerhalb einer Sammlung von AncestryDNA-Proben. Die Knoten in einem solchen Netzwerk sind die Proben und die Ränder zwischen Knoten sind die IBD-Verbindungen zwischen Proben. Wir beschreiben das Konzept des IBD-Netzwerks detailliert in Abschnitt 3. Bevölkerungsgruppen kann man sich als Teile des Netzwerks vorstellen, die eine hohe Konnektivität aufweisen – Knoten verfügen über eine höhere IBD-Rate (und längere IBD) mit anderen Knoten innerhalb einer gegebenen Bevölkerungsgruppe als mit Knoten außerhalb dieser. Siehe Abschnitt 4 für eine umfangreichere Diskussion von Bevölkerungsgruppen.
Für ein besseres Verständnis darüber, wieso wir Strukturen von Bevölkerungsgruppen in einem IBD-Netzwerk erwarten, untersuchen wir einige grundlegende Prinzipien der Populationsgenetik.
2.1 Genetische Bevölkerung
Wir beginnen mit einer Diskussion über das Konzept einer genetischen Bevölkerung. Es existiert eine Vielzahl von Definitionen in der Genetik-Literatur darüber, was eine Bevölkerung ausmacht. Wir definieren eine Bevölkerung als Gruppe von Menschen, die im Allgemeinen in unmittelbarer Nähe zueinander leben und über mehrere Generationen Nachwuchs erzeugen. Diese Definition ist bewusst vage im Bezug auf Größe und Umfang. Eine Bevölkerung kann eine große, lose verbundene Gruppe (z. B. alle Europäer) oder eine kleinere, enger verbundene Gruppe (z. B. die Iren) sein. Obwohl unsere Definition einer Bevölkerung beim Umfang vage bleibt, ist sie bei Zeit und Ort sehr spezifisch. Beispielsweise könnte eine Bevölkerung die Vorfahren aus Europa einschließen, die vor zehntausend Jahren lebten, während eine Andere die Menschen einschließen könnte, die vor 200 Jahren in Connecticut lebten.
Jede Bevölkerung verfügt über einen anderen Grad der genetischen Isolation. Wenn eine Bevölkerung über einen hohen Grad der genetischen Isolation verfügt, deutet dies darauf hin, dass Mitglieder der Bevölkerung selten Nachwuchs mit Personen außerhalb dieser Bevölkerung zeugen. Andererseits weist eine Bevölkerung mit niedrigem Isolationsgrad eine hohe Migration und Zumischung mit Bevölkerungen in der Umgebung auf. Über die Zeit hinweg entwickeln isolierte Bevölkerungen erkennbare Muster der genetischen Variation.
Neue Bevölkerungen können auf vielerlei Weisen erschaffen werden. Beispielsweise könnte eine kleine Gruppe von Personen aus einer historischen Population an einen neuen Ort migrieren und eine neue Bevölkerung erschaffen, die nicht länger Nachwuchs mit der ursprünglichen Bevölkerung zeugt. Es ist auch möglich, dass sich diese neue Bevölkerung von der historischen Bevölkerung abspaltet ohne den Ursprungsort zu verlassen. Eine weitere Möglichkeit ist es, dass mehrere Ursprungsbevölkerungen zusammen kommen und sich vermischen und so Nachwuchs mit gemischtem genetischen Material aus vorher getrennten Bevölkerungen zeugen. Bei all diesen Beispielen ist das gemeinsame Merkmal die Erschaffung einer Barriere des Genflusses, die zu einer Entwicklung erkennbarer Muster von genetischer Variation führen. Auf kleinster Ebene wirken viele Kräfte, wie Geografie, Krieg, Religion, Kultur, Politik und Ökonomie, die eine Partnerwahl beeinflussen können. Überraschend ist jedoch, dass diese persönlichen Entscheidungen einen signifikanten Einfluss auf den Fluss von genetischem Material über Zeit und Raum haben. Dies lässt folgende Frage zu: Können wir den Einfluss von Partnerwahlen durch unsere Vorfahren beobachten, indem wir unsere eigene DNA untersuchen? Wie unten gezeigt, ist das durchaus der Fall.
2.2 Ein anschauliches Beispiel
Nehmen wir ein einfaches Beispiel, das zeigt, wie genetische Isolation in einer Bevölkerung zu Strukturen in Bevölkerungsgruppen in einem IBD-Netzwerk führen kann.
In Abbildung 2.2 bilden wir eine Gründungspopulation mit zehn nicht-verwandten Personen aus zehn verschiedenen Bevölkerungen ab. Dabei gilt es zu beachten, dass die zehn Gründungsmitglieder keine langen IBD-Segmente in ihrer DNA teilen, da sie aus verschiedenen Bevölkerungen stammen. In diesem Beispiel bilden diese zehn Personen zufällige Paare und jedes der fünf Paare hat zwei Kinder, woraus eine zweite Generation aus zehn Personen entsteht. In dieser zweiten Generation sind nun einige Personen lose über IBD auf enger Familienebene verbunden.
Wir wiederholen dieses Experiment für zwei weitere Generationen: zehn nicht-verwandte Personen in der zweiten Generation wählen zufällig Partner aus und jedes Paar hat zwei Kinder, woraus eine dritte Generation aus 10 Personen entsteht. Zu guter Letzt formen zehn Personen aus der dritten Generation zufällige Paare und jedes Paar zeugt nur ein Kind, das zu einer vierten Generation mit fünf Personen führt.
Nach diesen drei Generationen der zufälligen Fortpflanzung teilen alle fünf Nachkommen in der vierten Generation wenigstens einen Teil ihres Erbguts mit jedem der anderen vier Cousins und Cousinen. Diese fünf Personen haben auch eine höhere IBD-Rate mit Personen in dieser neuen Bevölkerung als mit Menschen aus den ursprünglichen zehn Ahnenpopulationen. In einem IBD-Netzwerk mit Menschen aus vielen anderen Populationen würde diese Bevölkerung wahrscheinlich eine Bevölkerungsgruppe formen.
.jpg)
Obwohl dieses Beispiel einen sehr vereinfachten Fall darstellt, hilft er den Gedankengang hinter Populationen zu beschreiben und wie genetische Isolation Strukturen in Bevölkerungsgruppen in einem großen IBD-Netzwerk erzeugen kann. Natürlich bestehen echte Populationen aus hunderten oder tausenden Gründungsmitgliedern und sind im Allgemeinen nicht vollständig isoliert. Der Grad der Zumischung (die Wahl von Partnern von außerhalb der Population) und Migrationsbewegungen in einer Population beeinflussen die Stärke der Strukturen einer Bevölkerungsgruppe in einem IBD-Netzwerk.
Es ist ebenfalls wichtig zu beachten, dass das IBD-Netzwerk in unserem Beispiel vollständig in der vierten Generation verbunden ist und in großen Populationen findet man solche vollständigen Verbindungen nur selten. Es ist eher die höhere IBD-Rate unter Individuen der gleichen Population durch Eheschließungen von hunderten oder tausenden von Familien über mehrere Generationen hinweg, die eine modulare Struktur im Netzwerk erzeugt. Wenn dies auftritt, verfügen Personen über mehr IBD-Verbindungen zu anderen Personen der gleichen Bevölkerungsgruppe (oder Population), als sie es zu Personen aus anderen Bevölkerungsgruppen tun.
2.3 Beispiel einer modernen Bevölkerung in der Appalachenregion von West Virginia und in West Virginia
Wir schließen Abschnitt 2 mit der Diskussion über die Schaffung einer Population ab, die sich während des 18. Jahrhunderts im westlichen Virginia und West Virginia niedergelassen hat und nutzen diese, um die Basis unserer Bevölkerungsgruppen-Funktion zu bieten (Abbildung 2.3).
2.3.1 Die Geschichte der Besiedlung des westlichen Virginias und West Virginia
Die europäisch-amerikanische Besiedlung des westlichen Virginias und West Virginias begann in den 1730er Jahren, als die Virginia-Kolonie die Besiedlung seiner westlichen Berge bewarb, um einen Puffer zwischen ihren Städten und den Ureinwohnern zu schaffen (Rice 1993). Zwischen 1750 und 1780 wuchs die Gründungspopulation in dieser Region. Dies war eine Periode des Friedens, Wohlstands und der aggressiven Besiedlung des Shenandoah Valleys nach dem King George‘s War 1748 und einem Vertrag mit den Ureinwohnern im Jahr 1752. Die Briten verbaten eine weitere Besiedlung mit der Königlichen Proklamation von 1763, doch nach dem Amerikanischen Unabhängigkeitskrieg drangen die Amerikaner in das Territorium. Die Konstruktion von Straßen zwischen 1818 und 1846 führten zu einer weiteren Besiedlung und der Isolation von ländlichen Gebieten (Rice 1993). Daher war diese Region bis zur Mitte des 19. Jahrhunderts zum größten Teil eine ländliche, wachsende Population aus Siedlern mit britischem, deutschem oder schottisch-irischem Hintergrund.
Zwischen 1850 und 1890 erlebte diese Region eine Zeit der Industrialisierung und einen daraus folgenden Bevölkerungsboom, da sich die Kohleindustrie in West Virginia etablierte, die Entwicklung der C&O-Eisenbahnstrecke begann und Kohlestädte entlang der Strecke entstanden. Beispielsweise erlebte Kanawha County ein Bevölkerungswachstum von 700 % zwischen 1890 und 1910 (Laidley 1911 [310]). Mit dem Beginn des 20. Jahrhunderts wurde eine Änderung in diesem Muster deutlich, in Form einer massiven Emigration in die Industriestädte des Mittleren Westens und Westens im Zuge des Ersten Weltkriegs.
2.3.2 Diskussion über die Besiedlung des westlichen Virginias und West Virginias
In diesem Beispiel sehen wir die Erschaffung einer neuen Bevölkerung in der späteren Hälfte des 18. Jahrhunderts, die aus Gründungsmitgliedern mit schottisch-irischer, deutscher und britischer Herkunft besteht.
Auch wenn diese Population sicherlich DNA enthält, die zu ihrer entfernten schottisch-irischen, deutschen und britischen Herkunft zurückführt, hat die Verpaarung zwischen Gründern dieser neuen Population und ihren Nachfahren über viele Generationen zur Formation einer neuen Bevölkerung mit genetischer Variation geführt, die eine Verwandtschaft, wenn auch eine entfernte, mit ihrer Ursprungspopulation aufweist. Die Nachfahren dieser neuen Population sind Menschen, die große Mengen genetischen Materials mit vielen anderen Nachfahren dieser Population teilen. Familien haben durch die 1800er Jahre hinweg untereinander geheiratet, bis die Menschen nach dem Ersten Weltkrieg wegzogen. Doch sogar für die Nachfahren der Familien, die West Virginia vor einiger Zeit verließen, besteht die genetische Signatur in Form langer IBD-Segmente, die unter den Nachfahren trotz ihrer aktuelleren Familiengeschichte geteilt werden. Daher erwarten wir, diese Gruppe aus Nachfahren in unserer AncestryDNA-Datenbank zu finden, indem wir die IBD-Verbindungen zwischen diesen Personen verwenden. In den folgenden Abschnitten zeigen wir, wie wir diese und andere Generationen von Nachfahren in einem IBD-Netzwerk entdeckt haben.
Es ist wichtig zu beachten, dass die Beispiele in diesem Abschnitt als Beschreibung allgemeiner Prinzipien zur Motivation unseres Ansatzes der Verwendung von Gruppenerkennung ist, um Bevölkerungsgruppen in einem großen IBD-Netzwerk zu entdecken. Diese beiden Beispiele repräsentieren nicht die einzigartige Geschichte aller Populationen auf der Welt. Jede dieser Bevölkerungsgruppen, die wir entdecken, verfügen über ihre eigene, einzigartige Geschichte und Graden der genetischen Isolation sowie Migration. Dennoch sind einige der hier diskutierten Prinzipien auf viele Populationen anwendbar.
3. Erstellung eines IBD-Netzwerkes aus IBD-Verbindungen
In diesen und nachfolgenden Abschnitten stellen wir unsere verwendeten Methoden vor, um Bevölkerungsgruppen zu entdecken und zu annotieren.
Wir beginnen mit der Sammlung aller identifizierten, gepaarten IBD-Verbindungen zwischen AncestryDNA-Kunden. Ein Kundenpaar weist eine IBD-Verbindung auf, wenn es ein oder mehrere Segment(e) identischer DNA teilt. Die häufigste Erklärung für ein langes Segment identischer DNA bei zwei Individuen ist, dass diese Beiden einen einzelnen gemeinsamen Vorfahren teilen und daher IBD in den zwei Nachfahren auftritt.
.jpg)
Anhand des Beispiels von Kunde A haben wir durch den Vergleich seiner DNA mit denen anderer Kunden in unserer Datenbank, sieben andere Kunden identifiziert, die eine IBD-Verbindung zu ihm aufweisen (Kunden B, C, D, E, F, G und H). (Siehe Whitepaper zur Übereinstimmung für weitere Details zur Identifizierung von IBD-Verbindungen). Die genetischen Verbindungen in diesem kleinen Beispiel können visuell durch die Zeichnung von Rändern zwischen den identifizierten Paaren dargestellt werden, die eine Verbindung über ihre DNA vorweisen (Abbildung 3.1). In diesem speziellen Beispiel sind A, B, und C Cousins ersten Grades und somit alle drei über Ränder verbunden.
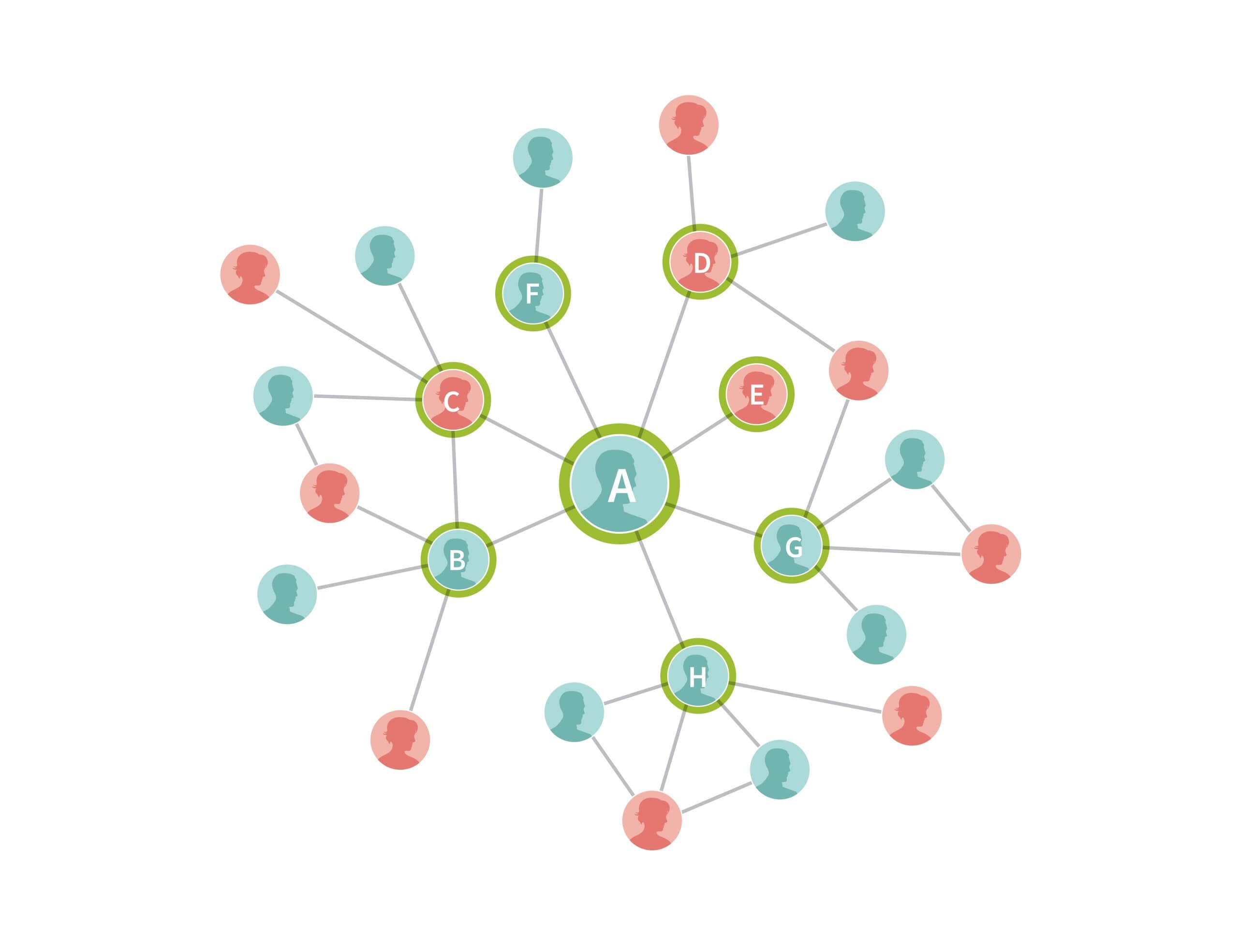
Als nächstes bauen wir auf diesem Beispiel auf, indem wir die IBD-Verbindungen einschließen, die wir für jeden der sieben Verbindungen von Kunde A gefunden haben (Abbildung 3.2). Die hinzugefügten Proben in diesem Schritt werden als grüne Kreise dargestellt. Die Größe des Netzwerks erweitert sich rapide mit dem Hinzufügen weiterer Menschen, indem wir die genetischen Verbindungen zwischen ihnen verfolgen. In einigen Fällen stehen diese neuen Proben also in Beziehung zueinander und in anderen Fällen sind diese Proben mit anderen verbunden, die bereits im Netzwerk eingeschlossen sind (die blauen und weißen Kreise). In beiden Fällen zeichnen wir Ränder, die die identifizierten IBD-Verbindungen zwischen Personen reflektieren.

Auf dieser Logik aufbauend, formen wir ein IBD-Netzwerk aus den IBD-Verbindungen, die wir unter den Millionen von Personen erkennen, die den AncestryDNA-Test durchgeführt haben. Natürlich wäre die Abbildung dieses Netzwerk in einem einzelnen Bild, wie oben, äußerst schwierig. Wir zeigen die IBD-Verbindungen zwischen einem Set aus 75 ausgewählten AncestryDNA-Proben (Abbildung 3.3), um zu veranschaulichen, wie ein kleiner Teil dieses Netzwerkes aussehen kann. Dies ist ein Beispiel einer gut vernetzten Gruppe aus Proben im AncestryDNA IBD-Netzwerk, und dennoch gibt es noch Personenpaare in dieser Gruppe für die wir noch keine IBD-Verbindung gefunden haben.
4. Netzwerk-Cluster durch Gruppenerkennung
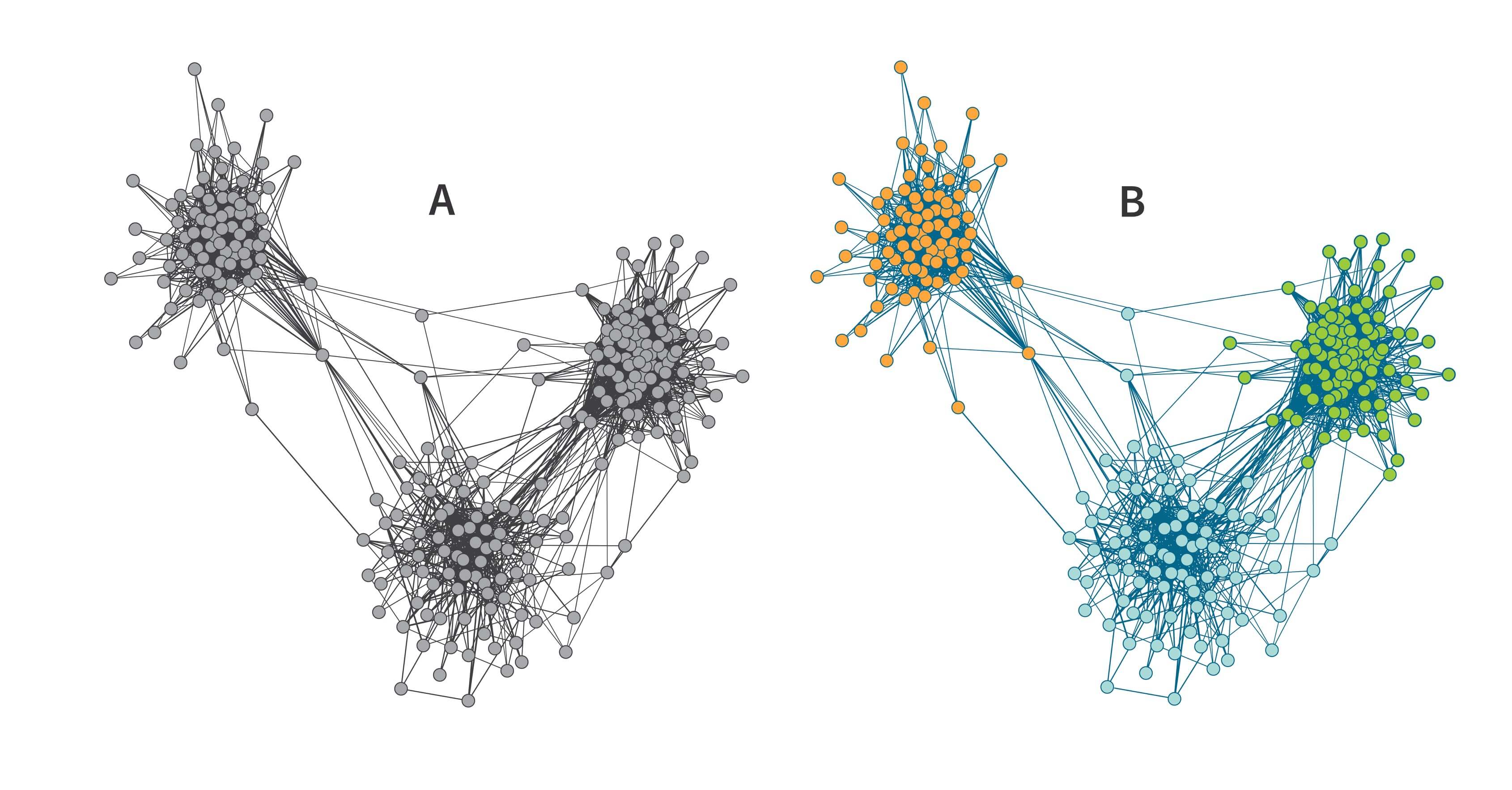
Wir können ein gegebenes IBD-Netzwerk in eng verknüpfte Gruppen unterteilen. Dazu verwenden wir die Louvain Methode – einen beliebten Gruppenerkennungsalgorithmus. Gruppenerkennungsalgorithmen sind Algorithmen zum Clustern von Netzwerken, die stark vernetzte Untergruppen eines Netzwerks identifizieren (Blondel et al. 2008, Csardi et al. 2008). Im Falle unseres IBD-Netzwerks repräsentieren diese Bevölkerungsgruppen Gruppen von Personen, die näher miteinander verwandt sind als mit anderen im Netzwerk.
In unserer visuellen Repräsentation des IBD-Netzwerks aus 75 Proben in Abbildung 3.3 ist kein klares Muster zu erkennen. In Abbildung 4.1 zeigen wir das gleiche Netzwerk mit neu ausgerichteten Knoten, um die Struktur des Netzwerks hervorzuheben. Genauer gesagt, haben wir die 75 Personen in drei Gruppen, Bevölkerungsgruppen, unterteilt, die wir dann als Bevölkerungsgruppe A (24 Personen, als grüne Kreise abgebildet), Bevölkerungsgruppe B (30 Personen, orange) und Bevölkerungsgruppe C (21 Personen, blau) bezeichnet haben. Es ist zu beachten, dass diese Bevölkerungsgruppen nicht visuell erkennbar sind, sondern durch einen Gruppenerkennungsalgorithmus, der auf dieses kleine IBD-Netzwerk angewandt wurde, der jeden Knoten zu einer Gemeinschaft zuweist.
| Bevölkerungsgruppe A (n=24) | Bevölkerungsgruppe B (n=30) | Bevölkerungsgruppe C (n=21) | |
|---|---|---|---|
| Bevölkerungsgruppe A | 276 (100 %) | 43 (6 %) | 9 (2 %) |
| Bevölkerungsgruppe B | 373 (86 %) | 163 (26 %) | |
| Bevölkerungsgruppe C | 185 (88 %) |
Der Gruppenerkennungsalgorithmus unterteilt intuitiv das Netzwerk in Untergruppen, die enger verbunden sind als das volle ursprüngliche Netzwerk. Wir können mittels einer Messungstechnik namens Netzwerkdichte messen, wie stark ein Netzwerk verbunden ist. Die Dichte ist die Anzahl der vorhandenen Ränder im Netzwerk geteilt durch die Anzahl der möglichen Ränder im Netzwerk. Im Falle des IBD-Netzwerks gilt, dass das Netzwerk maximal verknüpft ist, wenn es einen Rand zwischen jedem Personenpaar gibt. Nach der Gruppenerkennung in unserem obigen Beispiel sind die Personenpaare innerhalb der gleichen Bevölkerungsgruppe dichter miteinander verbunden als Individuen zwischen Gruppen. Beispielsweise sind 185 Ränder in Bevölkerungsgruppe C enthalten, für eine Dichte von 185 x 2 / (20 x 21) = 88 %, wohingegen nur 163 Ränder die Mitglieder in den Bevölkerungsgruppen B und C verbinden, für eine Dichte von 163 / (21 x 30) = 26 %.
Die Unterteilung dieses Netzwerks in drei Bevölkerungsgruppen verdeutlicht ein anderes wichtiges Konzept, das bei der Untersuchung von Mustern bei IBD-Verbindungen über viele Individuen in Betracht gezogen werden sollte: einige Personen haben die meisten oder alle IBD-Verbindungen innerhalb einer der Gruppen, wohingegen andere Personen über IBD-Verbindungen verfügen, die sich über mehrere Gruppen verteilen. Ein Beispiel für das Erstere ist ein Knoten in der unteren linken Ecke der Abbildung 4.1, die durch einen blauen Pfeil gekennzeichnet ist. Die Ränder dieser Knoten verbinden sich alle mit anderen Knoten innerhalb der gleichen Bevölkerungsgruppe (Bevölkerungsgruppe B). Dagegen zeigt der Pfeil in der Mitte der Abbildung eine Person, die zur Bevölkerungsgruppe B gehört, obwohl sie IBD-Verbindungen mit vielen Mitgliedern beider Bevölkerungsgruppen A und B, sowie einige mit Bevölkerungsgruppe C, aufweist. Daher ist die Stärke der Mitgliedschaft in einer bestimmten Gruppe für einige Personen größer als für andere.
Wir teilen das AncestryDNA IBD-Netzwerk mittels eines Ansatzes zur Gruppenerkennung in eng verknüpfte Untergruppen (Bevölkerungsgruppen). Durch die Anwendung eines schnellen Netzwerkgruppenerkennungsalgorithmus auf das IBD-Netzwerk, sind wir in der Lage Bevölkerungsstrukturen innerhalb dieses Netzwerks zu entdecken. In Abschnitt 6 diskutieren wir die Methode, wie wir die Gruppenerkennung rekursiv durchführen, um detaillierte Bevölkerungsstrukturen zu entdecken.
5. Interpretation der historischen und geografischen Charakteristiken von Bevölkerungsgruppen
Bevölkerungsgruppen werden exklusiv durch die IBD-Verbindungen zwischen Personen entdeckt. Wie in Abschnitt 2 beschrieben, erwarten wir, dass diese verknüpften Bevölkerungsgruppen jeweils eine Gruppe von Nachfahren einer bestimmten Population repräsentieren. Wie können wir jedoch die historische Population identifizieren, die für einen bestimmten Satz von Verbindungen verantwortlich ist? Dafür verlassen wir uns auf genetische Daten und Informationen, die in den Stammbäumen der Nachfahren von Bevölkerungsgruppen vorhanden sind. Da diese Verbindungen eine unweit zurückliegende gemeinsame Herkunft widerspiegeln, können wir nach gemeinsamen Merkmalen suchen, die von Personen in Bevölkerungsgruppen geteilt werden, um die genetischen Muster mit der jüngeren Geschichte in Beziehung zu setzen. Diese gemeinsamen Merkmale helfen es eine gemeinsame Zeit, einen Ort oder eine Herkunftspopulation aus den Nachfahren zu identifizieren. Zum Beispiel könnten die Menschen aus einer der Bevölkerungsgruppe Nachfahren irischer Immigranten sein, die während der Großen Hungersnot im 19. Jahrhundert in die Vereinigten Staaten auswanderten.
Für diese Analyse verwenden wir zwei Datensätze: (1) die Proportionen ethnischer Beimischungen in 26 globalen Populationen, die aus den Genotypen geschätzt werden (siehe Whitepaper: Einschätzung der genetischen Herkunft), und (2) Stammbäume, die von Benutzern gesammelt wurden, die den AncestryDNA-Test durchgeführt haben. Der Umfang und die Diversität dieser Daten erlaubt es uns, ein detailliertes historisches und geografisches Bild von der im IBD-Netzwerk entdeckten Bevölkerungsgruppe abzuleiten.
Bevor wir unseren Annotationsprozess unserer Bevölkerungsgruppen beschreiben, sollte beachtet werden, dass die Fähigkeit eine bestimmte Bevölkerungsgruppe derart zu annotieren stark von den verfügbaren Daten abhängig ist. Wenn beispielsweise keine Mitglieder einer Bevölkerungsgruppe Stammbäume erstellt haben, ist unsere Fähigkeit einen Herkunftsort zu identifizieren sehr eingeschränkt. Wichtig ist auch, dass eine Person nur mit einer bestimmten Bevölkerungsgruppe verbunden werden kann, wenn sie eine signifikante Menge genetischen Materials mit anderen Nachfahren aus der Bevölkerungsgruppe teilt. Ohne eine genetische Verbindung können wir Personen nicht unseren Bevölkerungsgruppen zuordnen. Allerdings hat das kontinuierliche Wachstum unserer AncestryDNA-Datenbank einen positiven Einfluss auf diese Einschränkungen.
5.1. Durchschnittliche Ethnizität
Das erste Merkmal, das wir bei jedem unserer Bevölkerungsgruppen betrachten, sind die aus der DNA geschätzten Anteile der genetischen Ethnizität. Diese auf Ethnizitäten basierten Annotationen können zur Schätzung verwendet werden, welche Vorfahrpopulationen unter den Personen einer Bevölkerungsgruppe über- oder unterrepräsentiert sind. In einigen Fällen können Bevölkerungsgruppen mit stark überrepräsentieren Vorfahrpopulationen zu bekannten Populationen in Beziehung gesetzt werden. Beispielsweise können Bevölkerungsgruppen, die relativ modernen Immigrantengruppen in der USA zugeordnet werden können, wie z. B. finnisch, jüdisch und irisch, aus den ethnisch-basierten Annotationen identifiziert werden. Auf der anderen Seite haben Bevölkerungsgruppen, die zu Gruppen aus New York State, Pennsylvania und Ohio zählen, ähnliche, nicht-unterscheidbare genetische Ethnizitätsprofile.
Abbildung 5.1 untersucht die genetischen Ethnizitätsprofile von Mitgliedern einer bestimmten Bevölkerungsgruppe, die durch das IDB-Netzwerk entdeckt wurden. Die durchschnittliche Ethnizität dieser Personen ist primär Irland zuzuordnen und deutet daher darauf hin, dass diese Personen irische Vorfahren besitzen.
.jpg.jpg)
5.2. Angereicherte Nachnamen
Als nächstes betrachten wir die Nachnamen von Vorfahren einer Bevölkerungsgruppe, indem wir gesammelte Stammbaumdaten verwenden. Wir sammeln alle Nachnamen der Vorfahren aus der jüngeren Vergangenheit, die den Personen einer Bevölkerungsgruppe zugeordnet wurden, um die Nachnamen für diese Bevölkerungsgruppen zusammenzufassen. Um Nachnamen hervorzuheben, die eine höhere Wahrscheinlichkeit besitzen ein Merkmal der Bevölkerungsgruppe zu sein und damit einen höheren informativen Wert über die historische oder demografische Signifikanz der Bevölkerungsgruppe bieten, quantifizieren wir die statistischen Beweise (mittels p-Wert), sodass jeder Nachname in einer gegebenen Bevölkerungsgruppe überrepräsentiert ist, im Vergleich zur Nachnamenverteilung aller Personen in einem vollem IDB-Netzwerk. Dann ordnen wir die Nachnamen nach den statischen Beweisen (kleinerer p-Wert) und wählen die zehn höchsten Nachnamen als die Nachnamen aus, die ein Merkmal der gegebenen Bevölkerungsgruppe darstellen. Zum Beispiel sind die am höchsten eingeordneten Nachnahmen aus den Annotationen Personen zugeordnet, die der irischen Bevölkerungsgruppe in Abbildung 5.1 angehören, einschließlich „McCarthy“, „Sullivan“, „Murphy“, „O‘Brien“, und „O‘Connor“ (siehe Abbildung 5.2).
.jpg)
5.3. Angereicherte Geburtsorte
Eine andere Annotationsart, die wir zur Kennzeichnung von Bevölkerungsgruppen verwenden, sind Geburtsorte von Vorfahren, die Personen einer Bevölkerungsgruppe zugeordnet wurden. Diese Orte bieten nützliche geografische Hinweise, die oftmals Bevölkerungsgruppen mit historischen Populationen in Verbindung bringen können. Bei dieser Analyse sammeln wir statistische Daten von Geburtsorten von Vorfahren einer Bevölkerungsgruppe über die Zeit hinweg und fassen diese Daten zusammen, damit wir sie geografisch visualisieren können. Dies erreichen wir, indem wir alle Geburtsorte innerhalb bestimmter Generationen auf die nächste Koordinate auf einem zweidimensionalen (2D) Rasterfeld konvertieren. Für jeden Rasterpunkt im 2D-Raster berechnen wir ein Chancenverhältnis (CV). Dieser CV sind die Chancen, dass ein gegebener Rasterpunkt in einem 2D-Raster, Mitgliedern der Bevölkerungsgruppe zugeordnet ist, geteilt durch die Chancen, dass der gleiche Rasterpunkt Benutzern zugeordnet ist, die keine Mitglieder der Bevölkerungsgruppe sind. Mittels dieser CV-Messung generieren wir eine Karte, die Rasterpunkte darstellt, dessen größtes Chancenverhältnis visuell über Bezeichnungen oder unterscheidbare Farben ausgedrückt wird. Auf diese Weise korrespondieren die hervorgehobenen Orte auf der Karte mit den geografischen Orten, die in einer gegebenen Bevölkerungsgruppe eine besondere Dichte aufweisen.
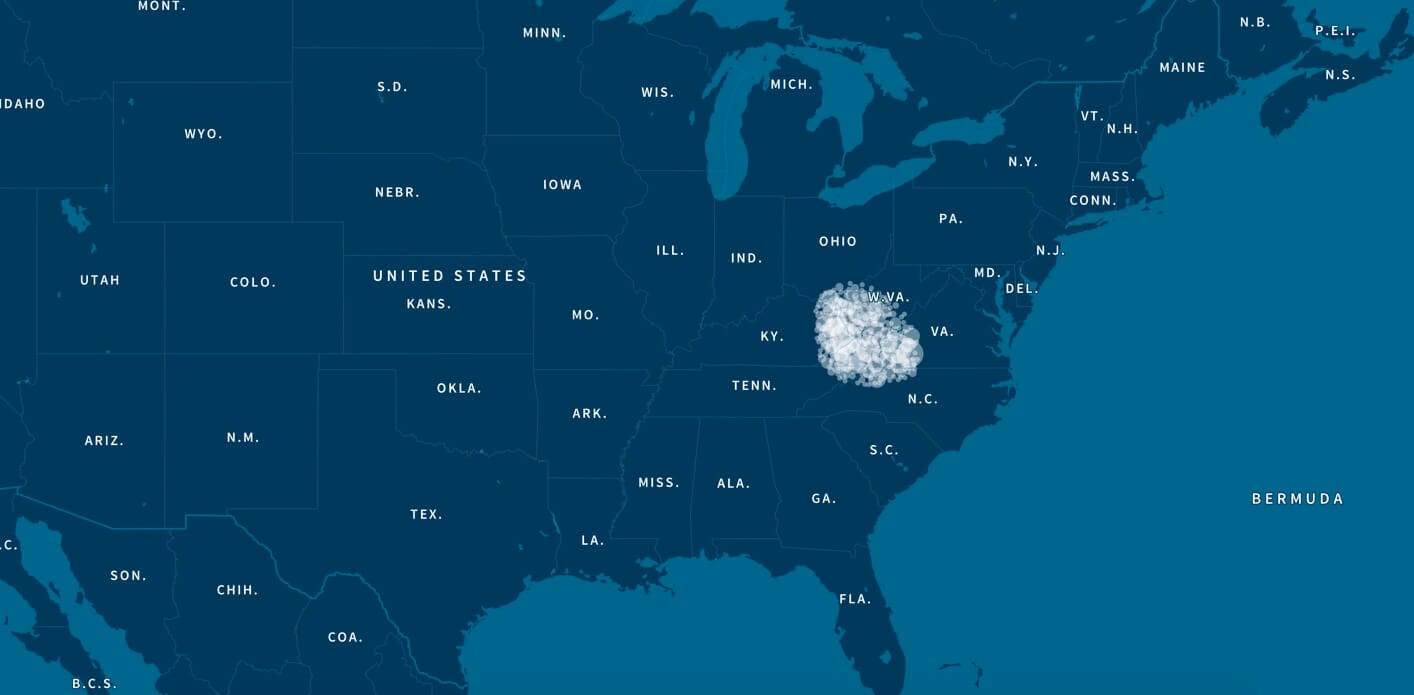
Beispielsweise zeigt Abbildung 5.3 angereicherte Geburtsorte von Vorfahren, die zwischen 1850 und 1910 geboren sind, und mit einer der irischen Bevölkerungsgruppen assoziiert sind. Diese Karte zeigt, dass Geburtsorte mit hohem CV (daher aussagekräftiger bzw. mehr angereichert) eine höhere Konzentration im südlichen Teil Irlands (Munster) aufweisen.
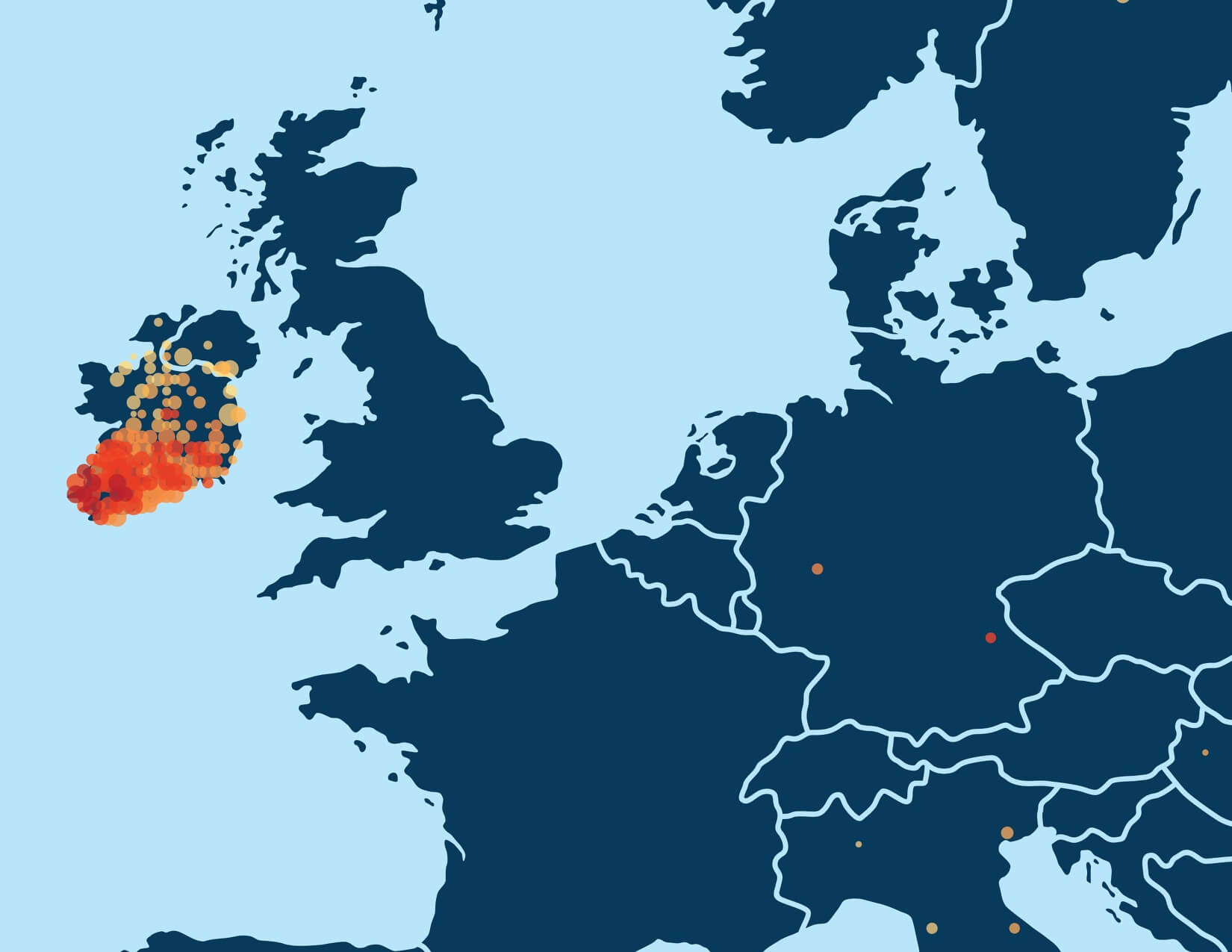
Zusätzlich zum Chancenverhältnis, ziehen wir auch das Verhältnis der Proben in Bevölkerungsgruppen in Betracht, die angestammte Geburtsorte in der für jede der Bevölkerungsgruppen identifizierten Region aufweisen. Dafür verwenden wir zunächst die Anreicherungspunkte der Geburtsorte, um Polygone um wichtige Orte zu erstellen, die spezifisch für jede der Bevölkerungsgruppen sind. (Diese Polygone werden auch im Produkterlebnis verwendet). Basierend auf diesen spezifischen Orten, können wir für jede Person einer Bevölkerungsgruppe festlegen, welche Vorfahren in dieser Region geboren wurden. In Abbildung 5.4, beispielsweise, zeigen wir das Verhältnis nach Generation von Vorfahren, die an Orten im Munster, Irland, Polygon geboren wurden. Für Personen, die dieser Bevölkerungsgruppe zugeordnet sind, gilt, dass 26,2 % ihrer Urgroßeltern innerhalb dieses Polygons geboren wurden. Für Personen, die dieser Bevölkerungsgruppe nicht zugeordnet sind, gilt, dass nur etwa 2 % ihrer Urgroßeltern an diesem gleichen Ort geboren wurden. Diese Analyse unterstützt unsere Interpretation, dass diese Bevölkerungsgruppe aus Nachfahren besteht, die in Munster, Irland lebten.
.jpg)
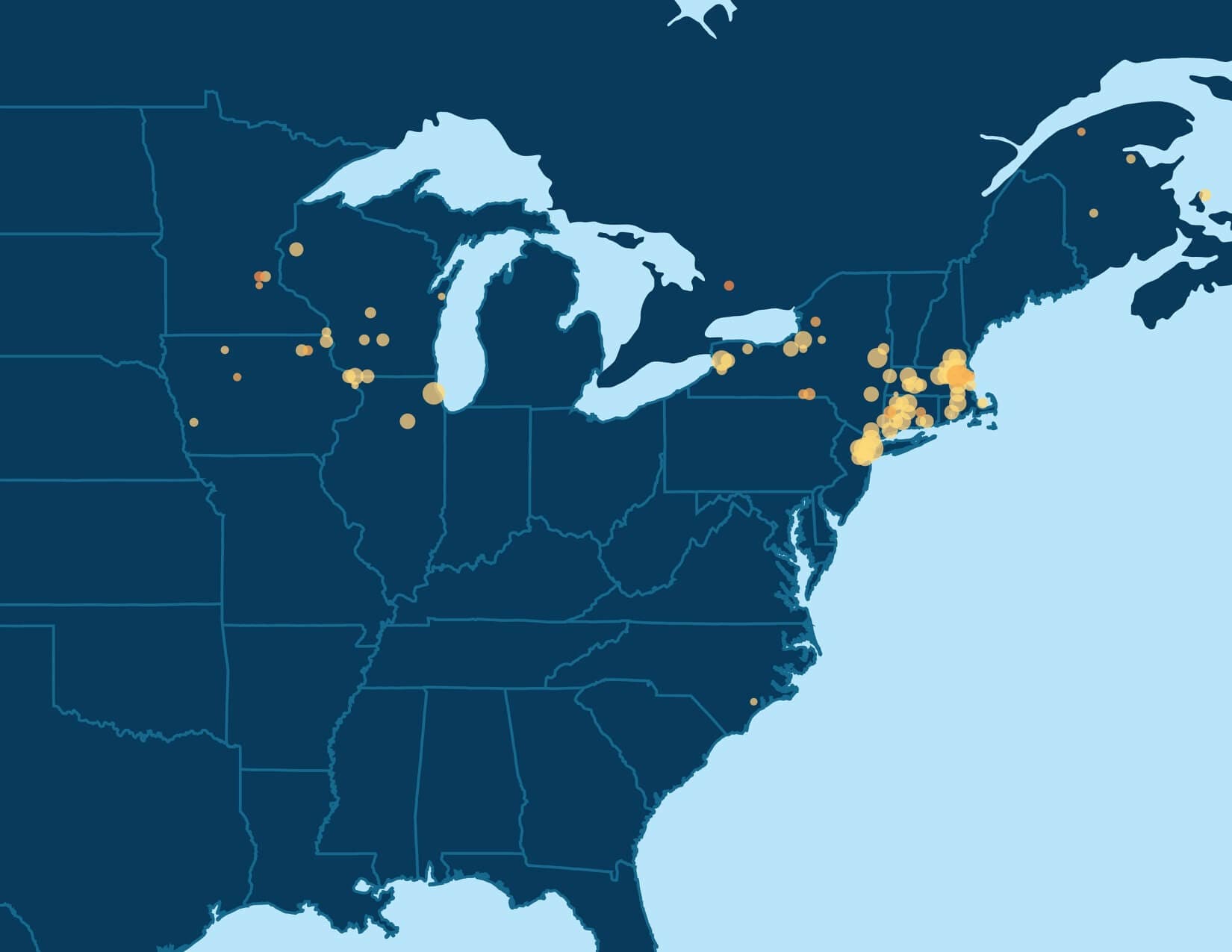
Wie vorher erwähnt, hängt unser Vertrauen in unsere Interpretation für jede Bevölkerungsgruppe von den Daten ab, die durch die Mitglieder Bevölkerungsgruppe zur Verfügung gestellt wurden. Um unsere Interpretationen für Bevölkerungsgruppen zu beurteilen, berücksichtigen wir die Anzahl der Annotationen, die Menschen in ihrem Stammbaum haben. Beispielsweise haben Personen in einigen Bevölkerungsgruppen weniger annotierte Vorfahren in den tieferen Ebenen ihres Stammbaums als andere. Zwei anschauliche Beispiele werden in Abbildung 5.5 gezeigt: Menschen aus dem westlichen Virginia neigen dazu mehr annotierte Vorfahren in tieferen Generationen zu besitzen als der Datenbankdurchschnitt, während Munster-Iren weniger annotierte Vorfahren in tieferen Generationen vorweisen. Daher können wir bei unserer Interpretation der Bevölkerungsgruppe aus dem westlichen Virginia sicherer sein, während wir bei der irischen Munster Bevölkerungsgruppe auf andere annotierte Daten zurückgreifen. Da viele Personen ihren Stammbaum nicht bis nach Munster, Irland zurückverfolgen können, betrachten wir die genetische Ethnizität. Wir haben herausgefunden, dass 98 % der Personen der Bevölkerungsgruppe >5 % irischer Herkunft sind und damit unsere Hypothese unterstützen, dass diese Mitglieder der Bevölkerungsgruppe aus Irland stammen.
.jpg)
5.4. Migrationsmuster
Letztlich untersuchen wir auch die Migrationsmuster von Vorfahren der Mitglieder von Bevölkerungsgruppen über die Zeit hinweg. Diese Muster erhalten wir aus den kumulierten Stammbaumdaten. Wir untersuchen, wie sich die Vorfahren von Menschen in der Bevölkerungsgruppe von einem Ort zum anderen bewegt haben, indem wir die Geburtsorte der Eltern und Kinder jeder Generation in allen Stammbäumen betrachten. Dadurch definieren wir einen Migrationspfad als Weg vom Geburtsort eines Elternteils zu einem Geburtsort eines Kindes.
Durch die Änderungen in diesen Migrationswegen erhalten wir oft weitere Einblicke in die Populationsdynamiken der Vorfahren dieser Bevölkerungsgruppe und wie sich diese Dynamiken über die Zeit verändert haben.
Wenn wir bspw. die irische Bevölkerungsgruppe aus Munster, Irland betrachten, sehen wir eine hohe Frequenz von Migrationswegen von Munster in die Vereinigten Staaten von 1825 bis 1875. Dieser Zeitrahmen entspricht der Migrationswelle von 6 Millionen Iren in die Vereinigten Staaten im 19. Jahrhundert, die seinen Höhepunkt 1852 während der Großen Hungersnot erreichte (Fitzgerald und Lampkin 2008 [8,181]).
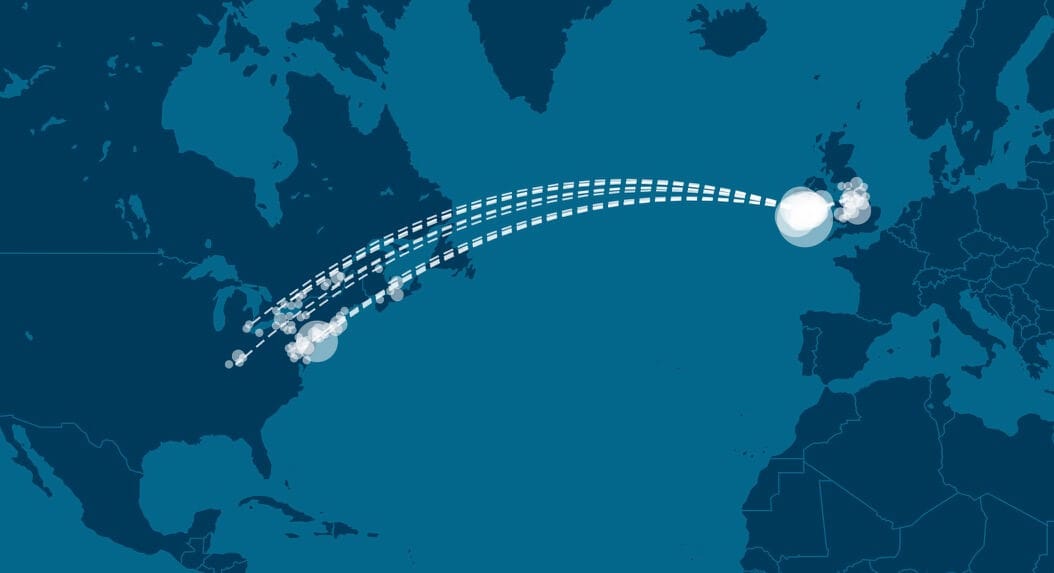
5.5 Interpretationen von Bevölkerungsgruppen
Auf Grundlage dieser vier Informationen – Ethnizität, Nachnamen, Geburtsorte und Migrationswege – können wir oft auf einen historischen Kontext schließen, der zu den starken genetischen Verbindungen zwischen Personen in der gleichen Bevölkerungsgruppe führt. Diese Interpretationen werden verwendet, um die Namen der Bevölkerungsgruppen in der Benutzererfahrung zu führen, sowie um präsentierte historische und andere Informationen zuzuordnen.
6. Rekursives Entdecken von detaillierten Bevölkerungsgruppen
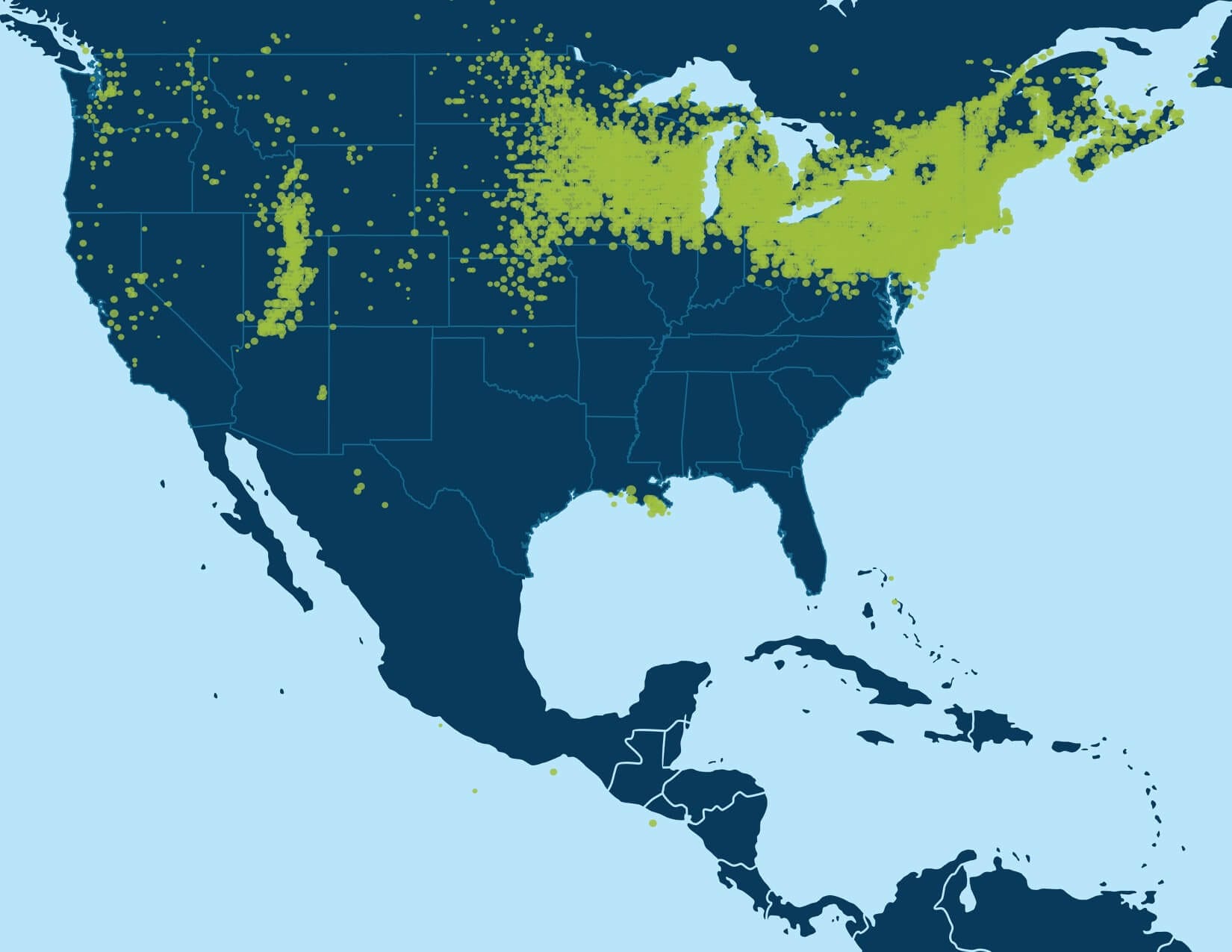
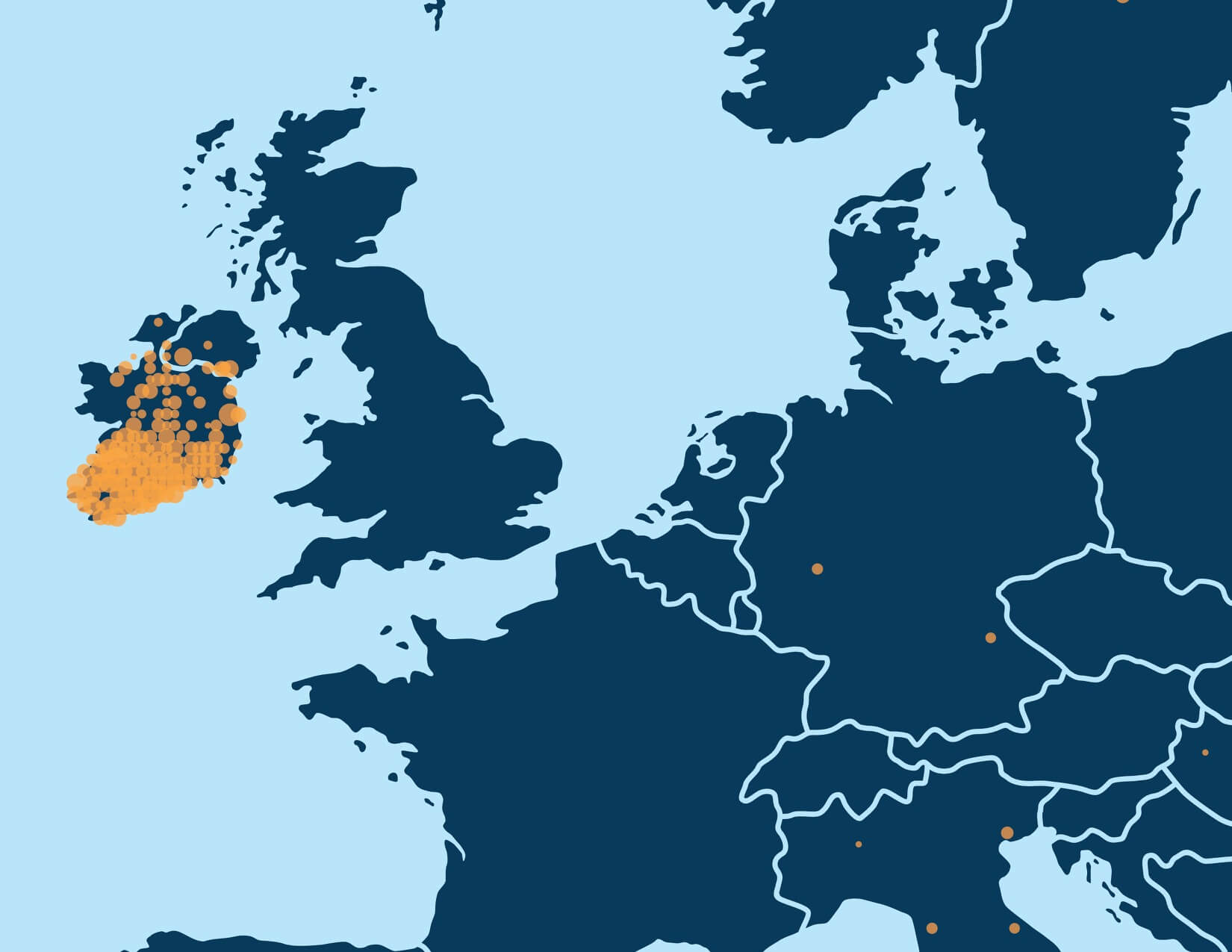
Bei Millionen von Proben in der AncestryDNA-Datenbank ermöglicht eine rekursive Ausführung der Gruppenerkennung die Identifizierung von detaillierten Strukturen im IBD-Netzwerk. Als wir das erste Mal die Gruppenerkennung auf das IBD-Probennetzwerk in der AncestryDNA-Datenbank angewandt haben, identifizierten wir zunächst eine Handvoll Bevölkerungsgruppen, die allgemein entweder leichte Genflussbarrieren repräsentieren, die hunderttausende Proben betreffen oder stärkere Genflussbarrieren, die kleinere Untergruppen im IBD-Netzwerk erschufen. Einige Beispiele von eher leichteren Genflussbarrieren schließen Bevölkerungsgruppen ein, die Menschen mit Vorfahren in den nördlichen USA und/oder Europa (Abbildung 6.1A) repräsentieren und Personen mit europäischer Herkunft, die Vorfahren in den südlichen USA aufweisen. Beispiele von Bevölkerungsgruppen aufgrund von stärkeren Genflussbarrieren schließen eine Person mit europäisch-jüdischer Abstammung und anderen Personen mit Abstammung aus Mexiko oder Lateinamerika ein (Abbildung 6.1B).
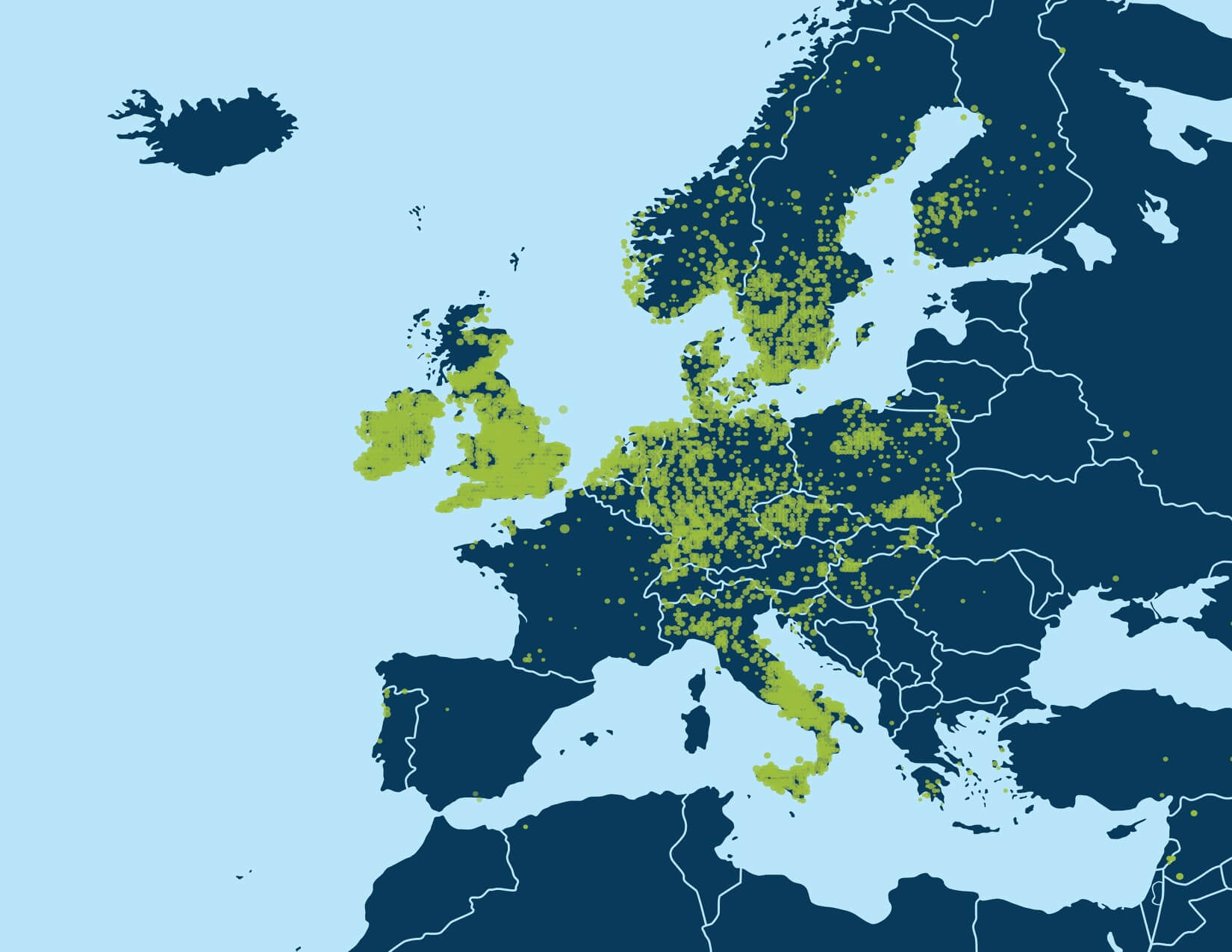
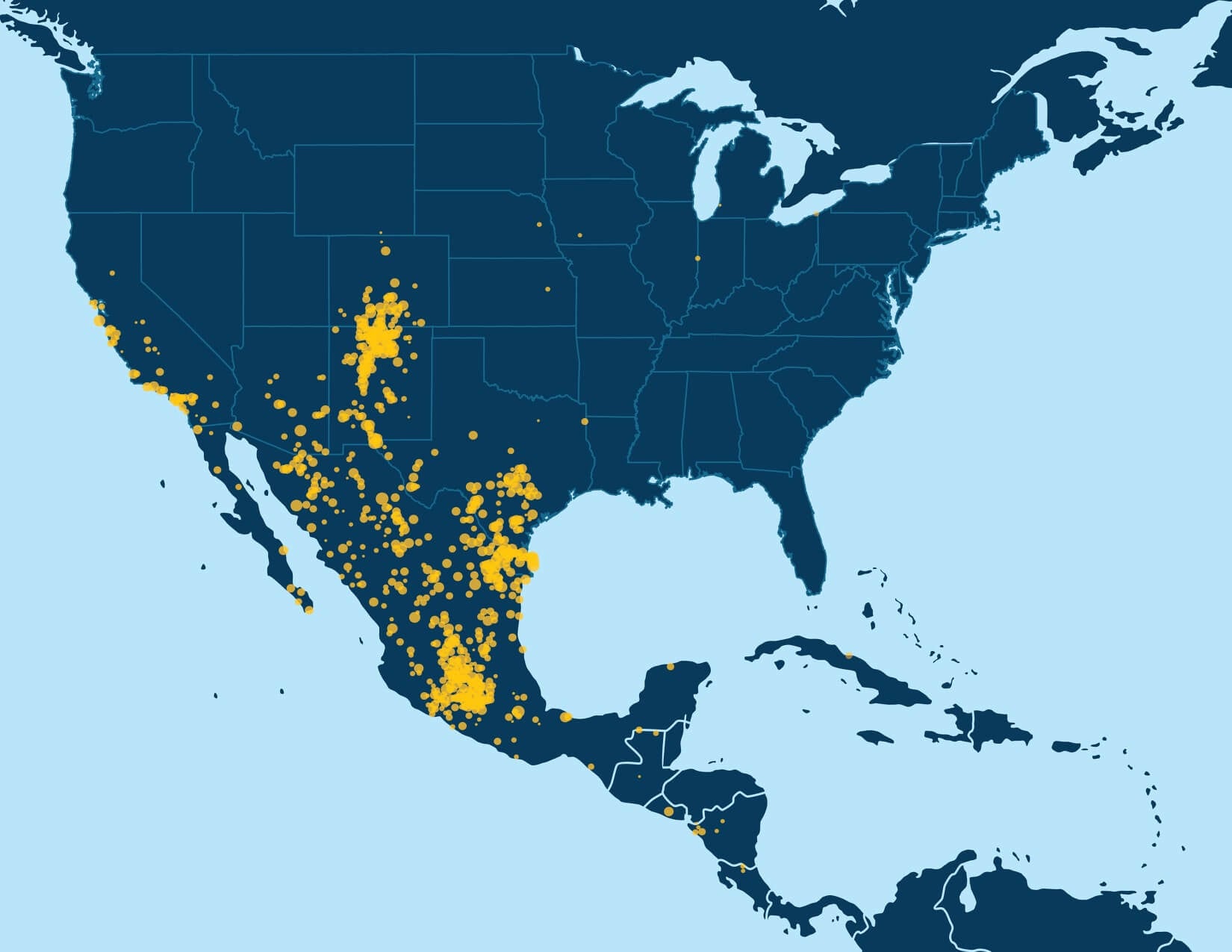
Eine wichtige Erkenntnis dieser Arbeit ist es, dass die Möglichkeit besteht, kleinere, höher aufgelöste Bevölkerungsgruppen durch die rekursive Anwendung des Gruppenerkennungsalgorithmus aufzudecken. Da jede der untersuchten Bevölkerungsgruppen selbst ein Netzwerk von IBD-Verbindungen darstellt, auf das wir den gleichen Gruppenerkennungsalgorithmus anwenden können, um Untergruppen zu entdecken, haben wir die Gruppenerkennung rekursiv durchgeführt. Unternetzwerke oder Bevölkerungsgruppen aus jeder Runde wurden rekursiv einer zusätzlichen Runde der Gruppenerkennung ausgesetzt, bis keine detaillierten Populationsstrukturen mehr zuverlässig erkannt werden konnten (Abbildung 6.2).
.jpg)
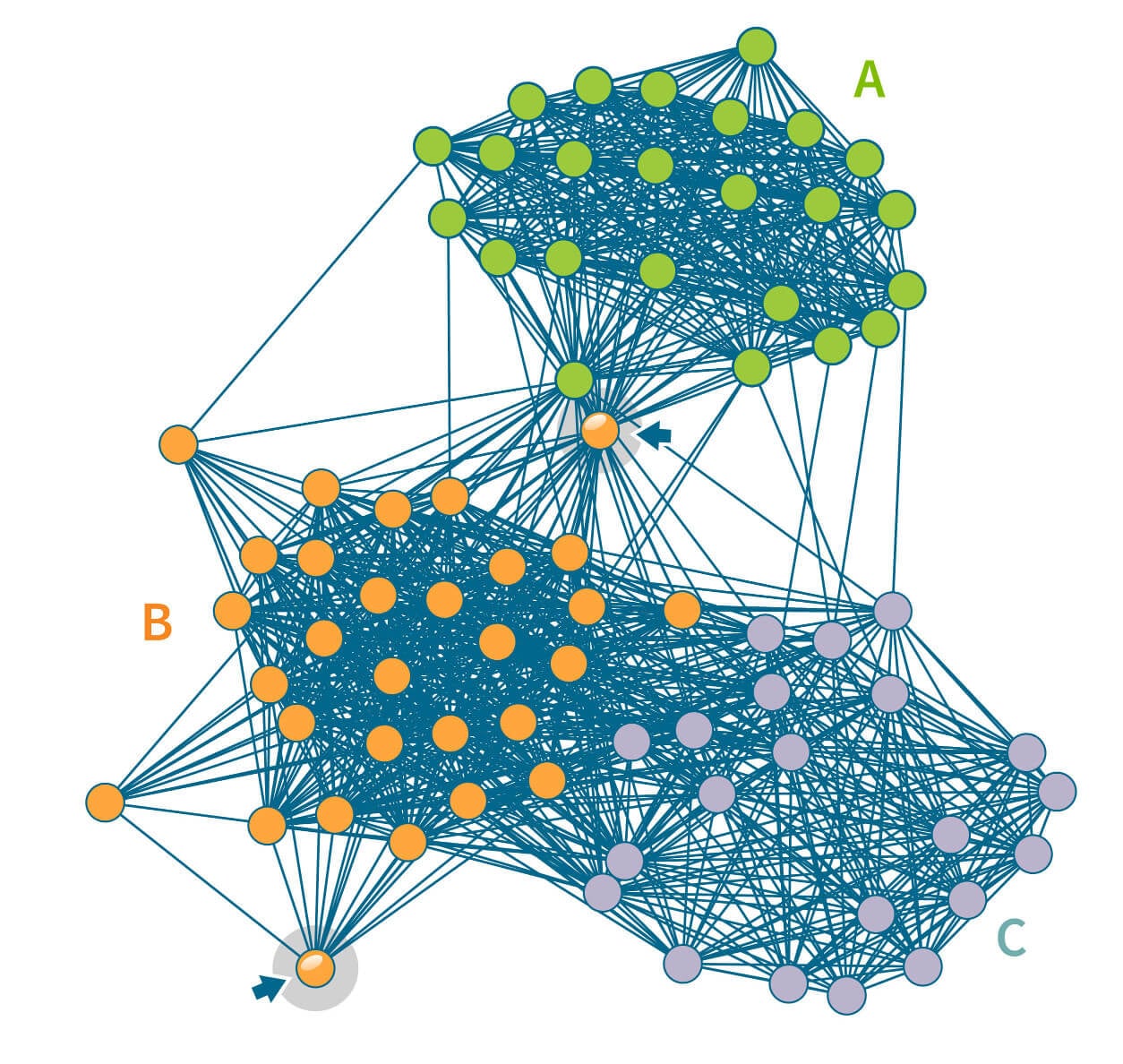
Zum Beispiel entdeckt die erste Runde der Gruppenerkennung eine große Bevölkerungsgruppe aus hunderttausenden Menschen mit Vorfahren in den nördlichen USA und/oder Europa (Abbildung 6.1A). Eine Ausführung der Gruppenerkennung nur auf diesem Unternetzwerk zeigt mehrere kleinere Bevölkerungsgruppen, die mit kleineren Populationsgruppen mit spezifischem Hintergrund übereinstimmen, wenn die annotierten Daten berücksichtigt werden. Wir sehen Bevölkerungsgruppen aus Menschen mit Vorfahren in Italien, Pennsylvania, New York oder das Vereinigte Königreich und Irland (Abbildung 6.3).
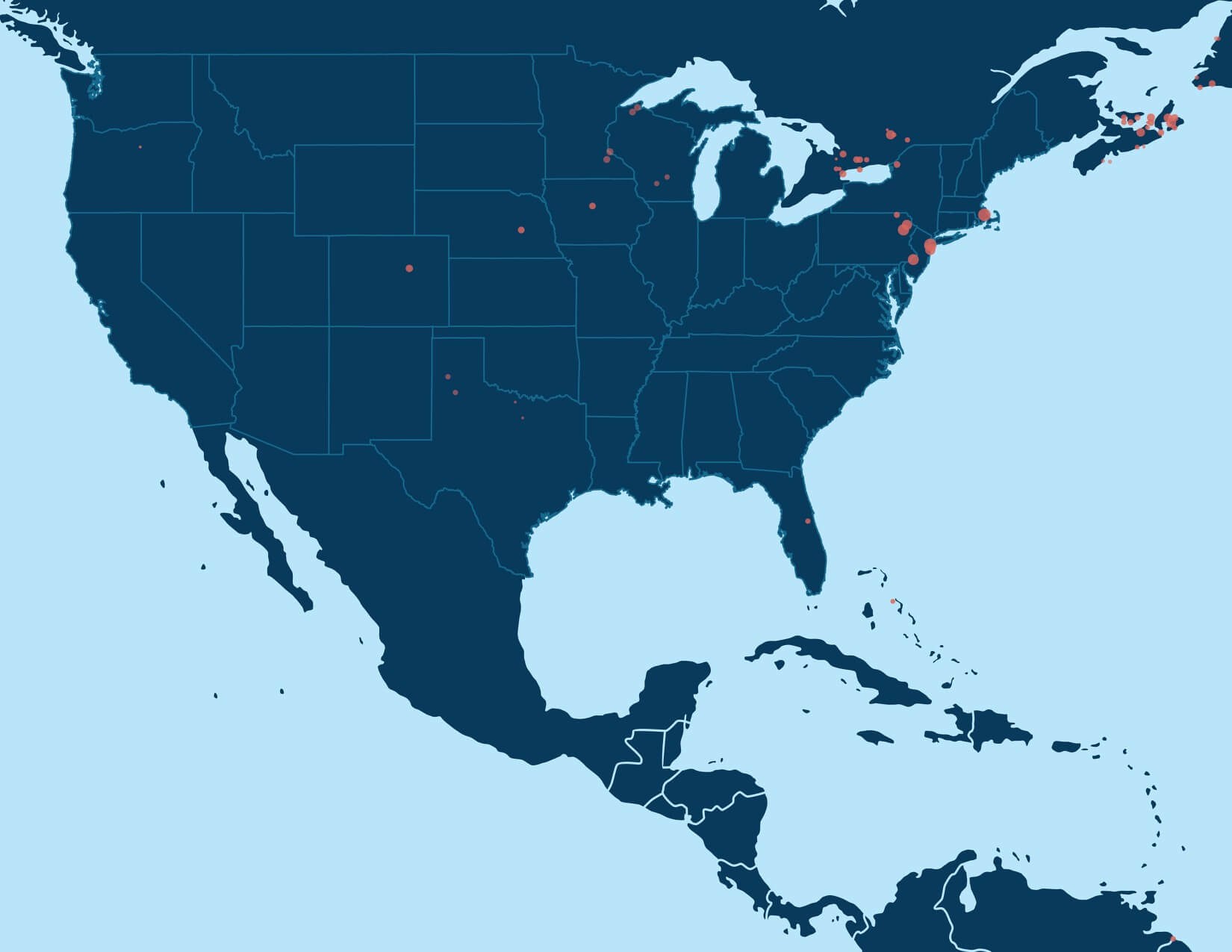
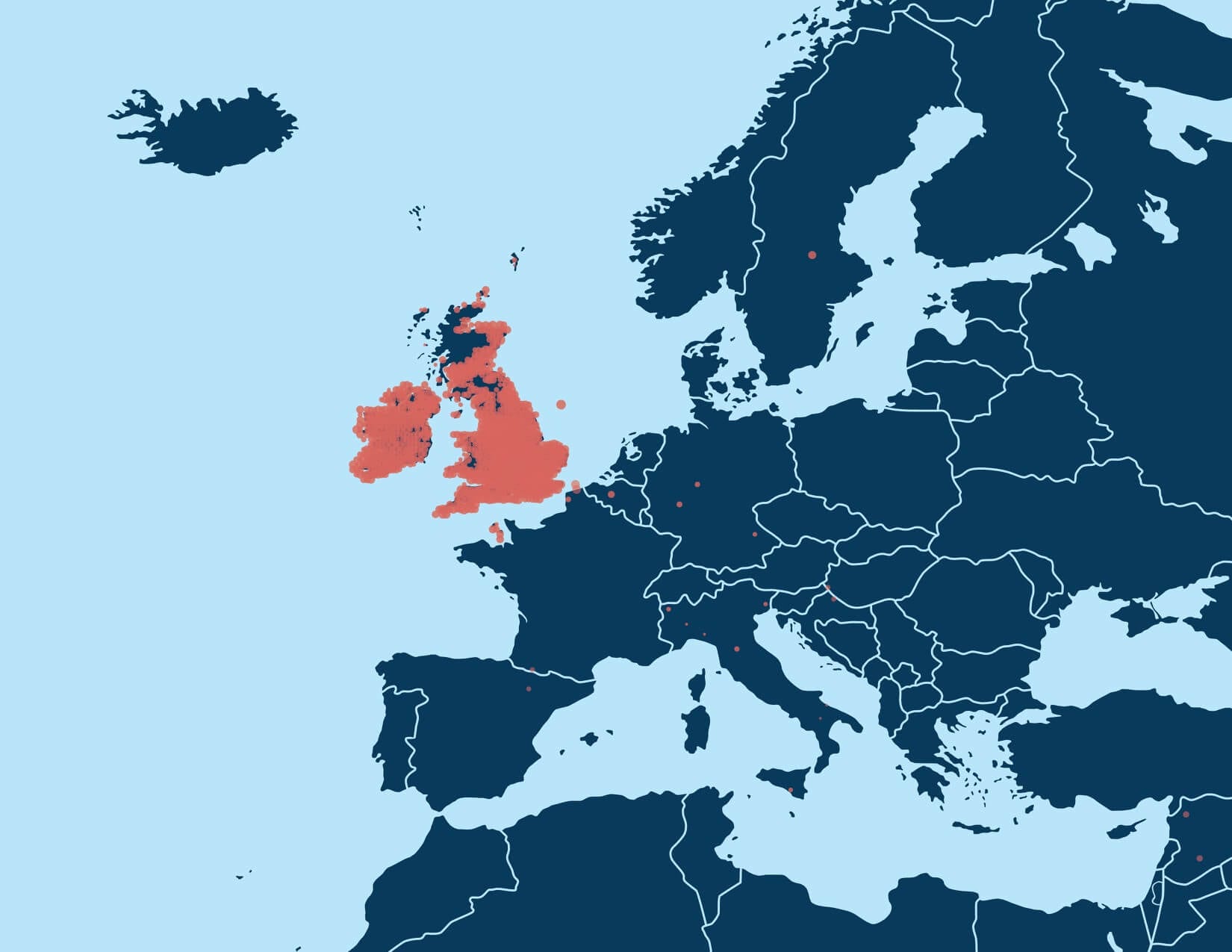
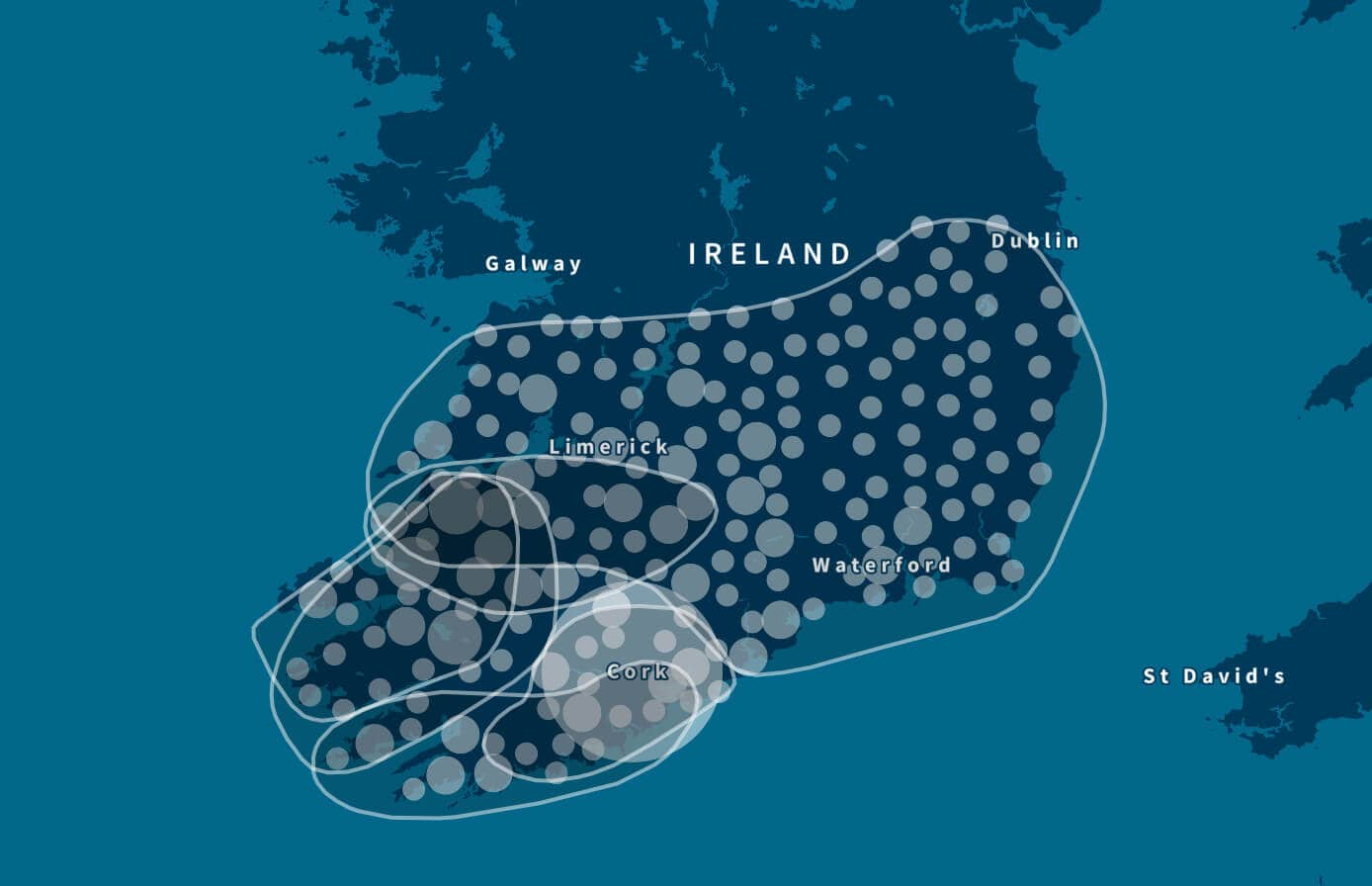
Die dargestellte Bevölkerungsgruppe in Abbildung 6.3 wurde mit dem gleichen Algorithmus entdeckt und repräsentiert eine detailreichere Populationsstruktur als die Bevölkerungsgruppen, die wir aus dem gesamten IBD-Netzwerk heraus entdeckt haben. Wir können die Gruppenerkennung noch einmal auf diesen kleineren Personenkreis anwenden. Wie zuvor, finden wir eine Reihe von Bevölkerungsgruppen, die jede mit noch detailreicheren Populationsstrukturen übereinstimmen. Wir finden drei Bevölkerungsgruppen, die Vorfahren aus Irland (Munster – siehe Abbildung 6.4, Ulster und Connacht) besitzen sowie Bevölkerungsgruppen in Neufundland, Nova Scotia und im Vereinigten Königreich.
Abermals können wir jede Bevölkerungsgruppe einzeln in Betracht ziehen und die Gruppenerkennung erneut ausführen. Durch die Ausführung der Gruppenerkennung auf die Bevölkerungsgruppe Munster, Irland, finden wir sechs Bevölkerungsgruppen, die mit mehreren (überschneidenden) Regionen in Munster übereinstimmen (Abbildung 6.5).
7. Zuweisung von Personen zu Bevölkerungsgruppen
Obwohl die Ergebnisse der rekursiven Applikation des Gruppenerkennungsalgorithmus auf das IBD-Netzwerk interessante detaillierte Bevölkerungsgruppen aufdeckt, benötigen wir dennoch eine Methode diese Einblicke unseren Kunden zu präsentieren. Eine Möglichkeit ist es, dass wir die einzelne Bevölkerungsgruppe auswählen, zu der jede Probe am Ende des Gruppenerkennungsalgorithmus zugeordnet wird und dann nur eine Bevölkerungsgruppenzuweisung anbieten. Allerdings unterläge dieser Ansatz zwei fundamentalen Einschränkungen. Erstens könnte jede einzelne AncestryDNA-Probe starke Verbindungen zu mehreren Bevölkerungsgruppen haben. Beispielsweise kann eine Person mit einer gemeinsamen Abstammung in der irischen und der italienischen Bevölkerungsgruppe eine starke Verbindung zu beiden haben, doch aufgrund der Art unseres verwendeten Gruppenerkennungsalgorithmus, zeigt das Endergebnis nur eine Zuweisung zu einer Bevölkerungsgruppe an. Zweitens ist die tägliche Ausführung der Gruppenerkennung für ein großes Netzwerk mit Millionen von Proben und Milliarden von Verbindungen rechnerisch unmöglich. Deswegen verwenden wir einen Algorithmus mit maschineller Lernfähigkeit, der beide Einschränkungen überwindet (Abb. 7.1).
.jpg)
Wir erzeugen ein Referenzpanel aus Proben für jede der Bevölkerungsgruppen, die wir während der rekursiven Gruppenerkennung entdecken, um Proben zu Bevölkerungsgruppen zuzuweisen. Jedes Referenzpanel wurde verfeinert, um Personen zu entfernen, die weniger der Bevölkerungsgruppe entsprechen und um enge Familienbeziehungen miteinzubeziehen. Für jedes Referenzpanel (repräsentierend für eine Bevölkerungsgruppe), das gewisse Qualitätsmetriken besteht, erstellen wir einen binären Klassifikator. Eine binäre Klassifikation ist ein Ansatz mit maschinellem Lernen, der eine Probe zu zwei Ergebnissen in gegebenen Merkmalen zuweist. Beispielsweise beschreiben gegebene Merkmale die IBD-Verbindung einer Probe zum Netzwerk, ein Klassifikator entscheidet dann entweder „Ja, zur Bevölkerungsgruppe zugewiesen“ oder „Nein, nicht zur Bevölkerungsgruppe zugewiesen.“ Da ein separater binärer Klassifikator für jede Bevölkerungsgruppe erstellt wird, besteht die Möglichkeit für eine Person für mehrere Bevölkerungsgruppen mit „Ja, zur Bevölkerungsgruppe zugewiesen“ klassifiziert zu werden, sofern sie Merkmale aufweist, die repräsentativ für diese Bevölkerungsgruppen sind. Zum Beispiel könnte eine Person mit gemeinsamer Abstammung aus zwei Bevölkerungsgruppen zu beiden zugewiesen werden. Dieser Ansatz der Zuweisung von Bevölkerungsgruppen kann als ein mehrwegiges Klassifikationsproblem beschrieben werden, in dem jede Probe als keine, eine oder mehrere Bevölkerungsgruppen klassifiziert werden könnte (Abbildung 7.2). Mittels dieses mehrwegigen Klassifikationsschemas sind wir in der Lage, Personen zu vielen Bevölkerungsgruppen zuzuweisen und die Tatsache zu umgehen, dass die Gruppenerkennung nicht mit jeder neuen Probe auf die vollständige AncestryDNA-Datenbank ausgeführt werden kann.
.jpg)
Die genutzten Merkmale in diesem Klassifikator werden durch die Zusammenfassung der IBD-Verbindungen aller Proben im IBD-Netzwerk und seine entdeckten Bevölkerungsgruppen herausgefunden. Da nicht jedes erstellte Merkmal für jeden Klassifikator nützlich ist, verwenden wir eine Auswahltechnik für Standardmerkmale, um nur die informationsreichsten Merkmale für jedes Modell auszuwählen. Die Anzahl der ausgewählten Merkmale variiert für jedes Klassifikationsmodell.
Bei jeder Bevölkerungsgruppe verwenden wir die ausgewählten Merkmale, um einen binären Klassifikator auszubilden, der gespeichert werden und bei der Zuweisung von AncestryDNA-Proben zu keiner, einer oder mehrerer relevanten Bevölkerungsgruppen verwendet werden kann. Wir verwenden einen Validierungsdatensatz (ein Datensatz aus Proben, die in eine Bevölkerungsgruppe gruppiert werden kann, wobei keine davon zur Ausbildung verwendet wird), um die Genauigkeit jedes Klassifikators einzuschätzen. Daher eignet sie sich für eine Klassifikation von „Ja, zur Bevölkerungsgruppe zugewiesen“, mit einem Konfidenzwert, den wir in die in Tabelle 7.1 gezeigten Kategorien einteilen.
| Konfidenzniveau | Genauigkeit des Validierungsdatensatzes |
|---|---|
| Sehr hohe Verbindung | Klassifikationen mit einer Genauigkeit im Validierungsdatensatz von etwa 95 % |
| Hohe Verbindung | Klassifikationen mit einer Genauigkeit im Validierungsdatensatz von etwa 80 % |
| Moderate Verbindung | Klassifikationen mit einer Genauigkeit im Validierungsdatensatz von etwa 60 % |
| Niedrige Verbindung | Klassifikationen mit einer Genauigkeit im Validierungsdatensatz von etwa 40 % |
| Sehr niedrige Verbindung | Klassifikationen mit einer Genauigkeit im Validierungsdatensatz von etwa 20 % |
Für eine einfache Darstellung werden diese Konfidenzniveaus zusammengefasst und Kunden in den folgenden drei Gruppen präsentiert:
- Sehr hohe Verbindung: Sehr wahrscheinlich
- Hohe und moderate Verbindung: Wahrscheinlich
- Niedrige oder sehr niedrige Verbindung: Möglich
Wir präsentieren dem Kunden im Produkterlebnis die Ergebnisse, die Sie heute sehen (Abbildung 7.3).
.jpg)
8. Fazit
In diesem Whitepaper beschreiben wir die Identifizierung und Zuordnung von Personen zu Bevölkerungsgruppen. Diese Bevölkerungsgruppen stimmen durch sehr aktuelle und manchmal dokumentierte historische Muster mit detaillierten Populationsstrukturen überein.
Zuerst identifizieren wir die genetischen Verbindungen, die durch einen gemeinsamen unweit zurückliegenden Vorfahren oder IBD unter Millionen von AncestryDNA-Proben definiert werden. Wenn diese Verbindungen in einem Netzwerk zusammengefasst wurden, zeigen unsere Berechnungsmethoden eng verknüpfte Cluster (Bevölkerungsgruppen), in denen Mitglieder jedes Clusters enger miteinander verwandt sind, als mit Mitgliedern anderer Bevölkerungsgruppen. Als nächstes annotieren wir diese Bevölkerungsgruppen mittels genetischen Ethnizitäten und benutzererzeugten Stammbäumen, um die vermutliche historische Herkunft solcher Populationsunterstrukturen zu identifizieren und, um temporale und geografische Migrations- und Besiedlungsmuster abzuleiten. Zuletzt leiten wir die Mitgliedschaft der AncestryDNA-Proben in diesen Bevölkerungsgruppen ab, indem wir maschinelle Lerntechniken anwenden und so einen detaillierten Bericht über ihre gegenwärtige Familiengeschichte in Nordamerika, Europa und anderswo erhalten.
Mit stetig wachsender AncestryDNA-Datenbank erwarten wir, dass sich unsere Fähigkeit zusätzliche Strukturen im IBD-Netzwerk zu entdecken weiter verbessert. Dies wird vermutlich zur Entdeckung von Bevölkerungsgruppen in neuen Gebieten der Welt führen und mit weitergehender Granularität, eine reichere Familiengeschichte für AncestryDNA-Kunden zur Folge haben.
9. Literaturverzeichnis
Blondel, Vincent D., Jean-Loup Guillaume, Renaud Lambiotte, und Etienne Lefebvre. „Fast Unfolding of Communities in Large Networks.“ Journal of Statistical Mechanics 2008, no. 10 (2008). doi:10.1088/1742-5468/2008/10/p10008.
Csárdi, Gábor und Tamás Nepusz. „The Igraph Software Package for Complex Network Research.“ InterJournal Complex Systems 1695 (2006).
Fitzgerald, Patrick und Brian Lambkin. Migration in Irish History, 1607–2007. Basingstoke: Palgrave Macmillan, 2008.
Laidley, W. S. History of Charleston and Kanawha County, West Virginia and Representative Citizens. Chicago: Richmond-Arnold Publishing, 1911.
Rice, Otis K. West Virginia: A History. Lexington, KY: University of Kentucky, 1985. Aufgerufen am 13. Mai 2015, https://muse.jhu.edu/.